توالی یابی چیست؟
توالی یابی DNA فرآیند تعیین توالی بازهای نوکلئوتیدی (As، Ts، Cs و Gs) در یک قطعه DNA است. امروزه با تجهیزات و مواد مناسب، تعیین توالی یک قطعه کوتاه DNA نسبتاً ساده است.
تعیین توالی کل ژنوم (همه DNA یک موجود زنده) یک کار پیچیده است. این امر مستلزم شکستن DNA ژنوم به قطعات کوچکتر، توالی یابی قطعات و مونتاژ توالی ها در یک “رشته” طویل است. با این حال، با ظهور روشهای جدیدی که در دو دهه گذشته توسعه یافتهاند، توالییابی ژنوم اکنون بسیار سریعتر و کمهزینهتر از پروژه ژنوم انسانی است.
کاربردهای توالی یابی
توالییابی DNA ممکن است برای تعیین توالی ژنهای منفرد، مناطق ژنتیکی بزرگتر (مثلاً خوشههایی از ژنها یا اپرونها)، کروموزومهای کامل یا کل ژنومهای هر موجود زنده مورد استفاده قرار گیرد. توالی یابی DNA همچنین کارآمدترین راه برای تعیین توالی غیرمستقیم RNA یا پروتئین ها (از طریق چارچوب های خواندن باز آنها) است. در واقع، توالی یابی DNA به یک فناوری کلیدی در بسیاری از زمینه های زیست شناسی و سایر علوم پزشکی مانند پزشکی قانونی و علم انسان شناسی تبدیل شده است.
توالی یابی سنگر: روش خاتمه زنجیره
مناطقی از DNA تا حدود 900 جفت باز به طور معمول با استفاده از روشی به نام توالی یابی Sanger یا روش خاتمه زنجیره ای توالی یابی می شوند.
توالی یابی سنگر برای اولین مرتبه توسط بیوشیمیدان بریتانیایی فردریک سنگر و همکارانش در سال 1977 توسعه یافت.
در پروژه ژنوم انسانی، توالی یابی سانگر برای تعیین توالی بسیاری از قطعات نسبتا کوچک DNA انسان استفاده شد. (این قطعات لزوماً 900 جفت باز یا کمتر نبودند، اما محققان توانستند در امتداد هر قطعه با استفاده از چندین دور توالی یابی سانگر “راه بروند”). قطعات بر اساس بخش های مشترک به هم متصل شدند تا توالی های مناطق بزرگتر DNA و در نهایت کل کروموزوم ها را تشکیل دهند.
اگرچه اکنون ژنومها معمولاً با استفاده از روشهای دیگری که سریعتر و کمهزینهتر هستند، توالییابی میشوند، توالییابی سانگر هنوز برای توالییابی قطعات منفرد DNA، مانند قطعات مورد استفاده در کلونینگ DNA یا تولید شده از طریق واکنش زنجیرهای پلیمراز (PCR) استفاده گستردهای دارد.
مواد لازم برای توالییابی سنگر
توالی یابی سنگر شامل ساختن کپی های زیادی از ناحیه DNA هدف است. مواد تشکیل دهنده آن شبیه به آنهایی است که برای تکثیر DNA در یک موجود زنده یا برای واکنش زنجیره ای پلیمراز (PCR) که DNA را در شرایط آزمایشگاهی کپی می کند، مورد نیاز است. آنها عبارتند از:
- یک آنزیم DNA پلیمراز
- یک پرایمر، که یک قطعه کوتاه از DNA تک رشته ای است که به DNA الگو متصل می شود و به عنوان یک “شروع کننده” برای پلیمراز عمل می کند.
- چهار نوکلئوتید DNA (dATP، dTTP، dCTP، Dgtp)
- DNA الگو که باید توالی یابی شود
با این حال، یک واکنش توالی یابی سانگر همچنین حاوی یک عنصر منحصر به فرد است:
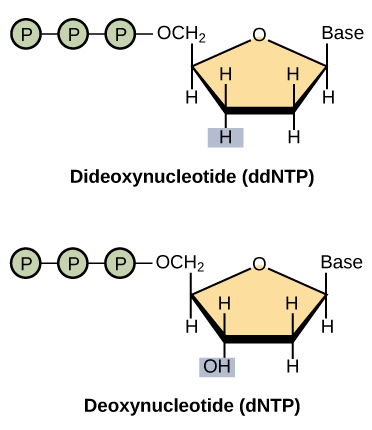
دی دئوکسی نوکلئوتیدها یا پایاندهنده زنجیرهای از هر چهار نوکلئوتید (ddATP، ddTTP، ddCTP، ddGTP)، که هر کدام با رنگ متفاوتی برچسبگذاری شدهاند.
نوکلئوتیدهای دی دئوکسی شبیه نوکلئوتیدهای معمولی یا دئوکسی اما با یک تفاوت کلیدی هستند: فاقد گروه هیدروکسیل در کربن 3 حلقه قند.
در یک نوکلئوتید معمولی، گروه هیدروکسیل 3 به عنوان یک “قلاب” عمل می کند و اجازه می دهد یک نوکلئوتید جدید به یک زنجیره موجود اضافه شود. هنگامی که دی دئوکسی نوکلئوتید به زنجیره اضافه شد، هیچ هیدروکسیل در دسترس نیست و هیچ نوکلئوتید دیگری نمی توان اضافه کرد. این زنجیره با دی اکسی نوکلئوتید خاتمه می یابد که با رنگ خاصی بسته به باز (A، T، C یا G) که حامل آن است مشخص می شود.
روش توالی یابی سنگر
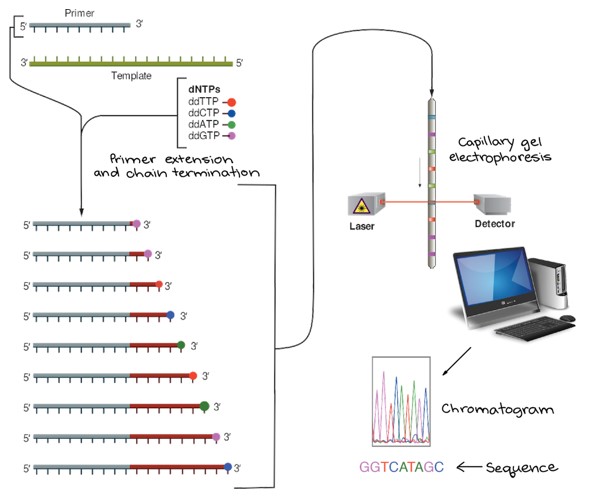
نمونه DNA برای تعیین توالی در یک لوله با پرایمر، DNA پلیمراز و نوکلئوتیدهای DNA (dATP، dTTP، dGTP و dCTP) ترکیب می شود. چهار دی دئوکسی نوکلئوتید پایاندهنده زنجیرهای که دارای رنگ است نیز اضافه میشوند، اما در مقادیر بسیار کمتر از نوکلئوتیدهای معمولی.
مخلوط ابتدا برای دناتوره کردن DNA الگو حرارت داده می شود (رشته ها را جدا می کند)، سپس خنک می شود تا پرایمر بتواند به الگوی تک رشته ای متصل شود. پس از اتصال پرایمر، دما مجدداً افزایش مییابد و به DNA پلیمراز اجازه میدهد تا DNA جدیدی را با شروع از پرایمر سنتز کند. DNA پلیمراز به افزودن نوکلئوتیدها به زنجیره ادامه می دهد تا زمانی که به جای یک نوکلئوتید معمولی، یک نوکلئوتید دی دئوکسی اضافه شود. در آن نقطه، هیچ نوکلئوتید دیگری نمی تواند اضافه شود، بنابراین رشته با دی دئوکسی نوکلئوتید به پایان می رسد.
این فرآیند در چند چرخه تکرار می شود و زنجیره گسترش می یابد تا زمانی که یک نوکلئوتید خاتمه به طور تصادفی ترکیب شود. یعنی لوله حاوی قطعاتی با طول های مختلف خواهد بود که هرکدام در یکی از موقعیت های نوکلئوتیدی در DNA اصلی یک دی دئوکسی نوکلئوتید قرار گرفته است (شکل زیر را ببینید). انتهای قطعات با رنگ هایی که نوکلئوتید نهایی آنها را نشان می دهد برچسب گذاری می شود.
پس از انجام واکنش، قطعات از طریق یک لوله بلند و نازک حاوی یک ماتریس ژل در فرآیندی به نام الکتروفورز ژل مویرگی عبور داده می شوند. قطعات کوتاه به سرعت در منافذ ژل حرکت می کنند، در حالی که قطعات بلند کندتر حرکت می کنند. همانطور که هر قطعه از “خط پایان” در انتهای لوله عبور می کند، توسط لیزر رنگ متصل شده به دی دئوکسی نوکلئوتیدها شناسایی میشود.
کوچکترین قطعه (که فقط یک نوکلئوتید بعد از پرایمر به پایان می رسد) ابتدا از خط پایان عبور می کند و سپس کوچکترین قطعه بعدی (پایان دادن به دو نوکلئوتید بعد از آغازگر) و غیره. بنابراین، از رنگهای فلوئوفورهایی که یکی پس از دیگری در آشکارساز ثبت میشوند، میتوان توالی قطعه اصلی DNA را شناسایی کرد. داده های ثبت شده توسط آشکارساز شامل یک سری پیک با شدت فلورسانس مختلف است، همانطور که در کروماتوگرام بالا نشان داده شده است. توالی DNA از پیک های کروماتوگرام خوانده می شود.
توالی یابی سنگر برای پروژه های در مقیاس بزرگتر، مانند توالی یابی کل ژنوم یا متاژنوم (“ژنوم جمعی” یک جامعه میکروبی) گران و ناکارآمد است. برای کارهایی از این قبیل، تکنیکهای جدید و در مقیاس بزرگ توالییابی سریعتر و کمهزینهتر هستند
توالی یابی نسل بعدی
این نام ممکن است شبیه پیشتازان فضا به نظر برسد، اما واقعاً این نام است! جدیدترین مجموعه فناوری های توالی یابی DNA در مجموع به عنوان توالی یابی نسل بعدی نامیده می شود.
انواع تکنیک های نسل بعدی توالی یابی وجود دارد که از فناوری های مختلفی استفاده می کنند. با این حال، اکثر آنها مجموعه ای از ویژگی های مشترکی دارند که آنها را از توالی سنگر متمایز می کند:
- توالی یابی قطعات بزرگی از DNA را به صورت موازی انبوه انجام می دهد، که در مقایسه با توالی یابی Sanger مبتنی بر الکتروفورز مویرگی مزایایی را در توان عملیاتی و مقیاس ارائه می دهد.
- وضوح بالایی برای به دست آوردن نمای باز به باز از یک ژن، اگزوم یا ژنوم ارائه می دهد.
- اندازه گیری های کمی را بر اساس شدت سیگنال ارائه می دهد.
- تقریباً تمام انواع تغییرات DNA ژنومی، از جمله انواع تک نوکلئوتیدی، درجها و حذفها، تغییرات تعداد کپی و ناهنجاریهای کروموزومی را تشخیص میدهد.
- توان عملیاتی و انعطاف پذیری بالایی را برای مقیاس بندی مطالعات و توالی چندین نمونه به طور همزمان ارائه می دهد
روش های متداول توالییابی DNA
- Whole-Genome Sequencing
توالی یابی کل ژنوم جامع ترین روش برای تجزیه و تحلیل ژنوم است. کاهش سریع هزینه های توالی یابی و توانایی به دست آوردن اطلاعات ارزشمند در مورد کل کد ژنتیکی، این روش را به یک ابزار تحقیقاتی قدرتمند تبدیل کرده است.
- Targeted Resequencing
با توالی یابی مجدد هدفمند، زیرمجموعه ای از ژن ها یا مناطق ژنوم جداسازی و توالی یابی می شوند و به دانشمندان این امکان را می دهد که زمان، هزینه ها و تجزیه و تحلیل را بر روی مناطق خاص مورد نظر متمرکز کنند.
- Methylation Sequencing
با استفاده از قدرت NGS، هم تجزیه و تحلیل گسترده ژنوم و هم رویکردهای هدفمند میتوانند بینشی در مورد الگوهای متیلاسیون DNA در یک سطح نوکلئوتیدی به محققان ارائه دهند.
- ChIP Sequencing
با ترکیب سنجش رسوب گذاری ایمنی کروماتین (ChIP) و توالی یابی تراشه (ChIP-Seq) یک روش قدرتمند برای شناسایی مکان های اتصال DNA در سطح ژنوم برای فاکتورهای رونویسی و سایر پروتئین ها است.